Nach dem Vorbild der Natur
13.07.2021, 07:35 Uhr
Mit KI zu neuen Arzneistoffen
KI kann gezielt die biologische Aktivität von Naturstoffen erkennen, wie Forschende der ETH Zürich gezeigt haben. Darüber hinaus hilft die Technologie, Moleküle zu finden, welche die gleiche Wirkung wie ein Naturstoff haben, aber einfacher in der Herstellung sind.
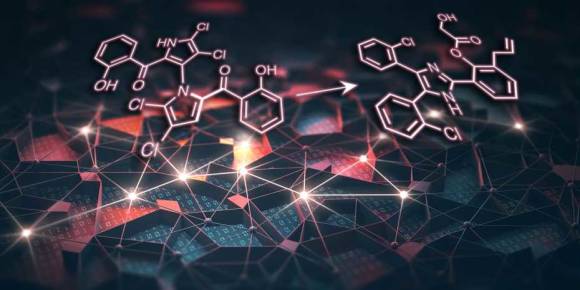
Das von einer künstlichen Intelligenz entworfene Molekül rechts hat dieselben pharmakologischen Eigenschaften wie der Naturstoff Marinopyrrol A (links)
(Quelle: Colourbox / Montage ETH Zürich)
Die Natur hält ein riesiges Reservoir an Heilmitteln bereit. «Über 50 Prozent aller heutigen Medikamente sind von der Natur inspiriert», sagt Gisbert Schneider. Dennoch ist der Professor für Computer-gestützte Medikamentenentwicklung an der ETH Zürich überzeugt, dass bis anhin erst ein kleiner Teil des Potenzials der Naturstoffe genutzt wird. Zusammen mit seinem Team konnte er nun zeigen, wie mit Methoden der künstlichen Intelligenz (KI) gezielt neue Arzneianwendungen von Naturstoffen gefunden werden können. Ausserdem lassen sich mit KI-Methoden auch Alternativen zu den Naturstoffen finden, die gleich wirken, aber wesentlich einfacher und damit günstiger in der Herstellung sind.
Zielmoleküle von Naturstoffen
Damit ermöglichen die ETH-Forschenden die Weiterentwicklung der Medizin: Insgesamt verfügen wir heute nämlich erst über rund 4000 grundsätzlich unterschiedliche Arzneistoffe. Diesen stehen je nach Schätzung bis zu 400'000 unterschiedliche menschliche Proteine gegenüber, von denen jedes ein Ziel für ein Medikament sein könnte. Dass sich Schneider bei der Suche nach neuen Arzneistoffen auf die Natur fokussiert, hat gute Gründe. «Die meisten Naturstoffe sind per Definition potenzielle Wirkstoffe, die über evolutionäre Mechanismen ausgewählt wurden», sagt er.
Während Wissenschaftler früher auf der Suche nach neuen Wirkstoffen ganze Naturstoffsammlungen durchforsteten, haben Schneider und sein Team den Spiess nun umgedreht: Sie suchen zunächst mithilfe des Computers nach möglichen Zielmolekülen von Naturstoffen und können auf diese Weise die pharmakologisch relevanten Verbindungen identifizieren. «Die Chancen, auf diesem Weg medizinisch bedeutsame Wirkstoff-Zielprotein-Paare zu finden, sind deutlich grösser als mit dem herkömmlichen Screening», betont Schneider.
Mit bakteriellem Molekül getestet
Getestet haben die ETH-Chemiker ihr Konzept mit Marinopyrrol A, einem bakteriellen Molekül, von dem bekannt ist, dass es antibiotisch und entzündungshemmend wirkt sowie anti-Krebs-Eigenschaften aufweist. Mit welchen Proteinen im menschlichen Körper der Naturstoff dabei wechselwirkt, war bis anhin jedoch weitgehend unerforscht.
Um mögliche Zielproteine von Marinopyrrol A zu finden, verwendeten die Forschenden einen von ihnen entwickelten Algorithmus. Dieser verglich mit maschinellen Lern-Modellen die pharmakologisch interessanten Teile von Marinopyrrol A mit den entsprechenden Mustern von Wirkstoffen, von denen bekannt ist, an welche Zielproteine sie sich heften. Anhand der Musterübereinstimmungen konnten die Forschenden acht menschliche Rezeptoren und Enzyme identifizieren, an die sich das bakterielle Molekül heften könnte. Diese sind unter anderem an Entzündungs- und Schmerzprozessen sowie am Immunsystem beteiligt.
Laborexperimente bestätigten, dass Marinopyrrol A tatsächlich mit den meisten der vorhergesagten Proteine messbare Wechselwirkungen eingeht. «Unsere KI-Methode kann die Proteinziele von Naturstoffen mit einer Zuverlässigkeit von oftmals über 50 Prozent eingrenzen und vereinfacht damit die Suche nach neuen Arzneistoffen», hält Schneider fest.
Autor(in)
Daniel
Meierhans, ETH-News